CRISPR/Cas
Referência : Ferraz, R., Coimbra, S., Correia, S., Canhoto, J., (2023) CRISPR/Cas, Rev. Ciência Elem., V11(3):009
Autores: Ricardo Ferraz, Sílvia Coimbra, Sandra Correia, Jorge Canhoto
Editor: João Nuno Tavares
DOI: [https://doi.org/10.24927/rce2023.009]

[editar] Resumo
As técnicas de modificação de genomas têm como objetivo a alteração de uma sequência específica de DNA. Com o evoluir destas técnicas foi possível desenvolver sistemas que envolvem enzimas reprogramáveis que estão no cerne das técnicas de edição genética atuais. Entre estas novas abordagens, um novo sistema viria revolucionar o mundo da engenharia genética, a técnica CRISPR/Cas. Esta técnica apresenta diversas aplicações na edição de genomas de espécies vegetais, tendo sido utilizada para melhorar diversas características, como a produtividade e a gestão de stresse, em plantas com interesse económico como o tomateiro. Embora esta nova abordagem apresente alguns pormenores que podem ainda ser melhorados, a potencialidade que apresenta para a modificação de genomas torná-la-á muito importante no melhoramento de plantas num futuro próximo.
As tecnologias de modificação de genomas têm como objetivo a alteração de uma sequência específica de DNA. A edição genética com fins biotecnológicos começou a ser aplicada nos anos 70 do século XX, com o estudo da recombinação homóloga, processo no qual uma sequência de DNA ladeada por regiões homólogas do DNA hospedeiro pode ser integrada no genoma deste[1]. No entanto, a utilização direta desta técnica apresenta várias limitações, como a baixa eficácia de recombinação e a integração aleatória em zonas do genoma não desejadas[2]. Para além disso, embora a utilização de organismos geneticamente modificados tenha um forte e positivo impacto no melhoramento de espécies vegetais com interesse económico, estas tecnologias não têm uma aceitação pública generalizada, em especial nos países europeus, o que tem dificultado a sua adoção[3], [4]. A aplicação da recombinação homóloga no melhoramento de plantas envolve a adição de material genético numa planta de interesse, modificando assim o seu genoma, sendo que o material genético adicionado pode ser da mesma planta ou ser estranho à planta a ser modificada, que, neste último caso, passa a ser designada de planta transgénica[5]. A baixa frequência de integração num local específico desejado e a necessidade de utilizar genes marcadores e/ou de seleção limitam bastante a utilização das técnicas de recombinação genética[6].
Com a evolução das técnicas de edição genética, foi possível desenvolver novos sistemas que não envolvem as antigas enzimas de recombinação, mas sim novas enzimas de restrição e nucleases reprogramáveis que estão no cerne das técnicas de edição genética atuais[7]. Para além disso, foi também crucial a associação entre as quebras na dupla cadeia do DNA (Double-Strand Breaks, DSB) e uma maior integração de DNA estranho no material genético de células de mamíferos[8], [9]. A descoberta de meganucleases, enzimas de restrição com sequências de reconhecimento de 20 a 30 pares de bases (pb) envolvidas na duplicação de genes, deu-se em 1985[10], mas foi nove anos mais tarde, com a descoberta das DSB, que estas enzimas foram associadas pela primeira vez à edição genética[11]. Após DSB, a maquinaria de reparação do DNA da célula repara a quebra: ou por ligação de extremidade não homóloga (Non-Homologous End Joining, NHEJ), inserindo ou retirando nucleótidos, o que pode levar a um knock-out do gene onde se deu a quebra; ou por reparação direcionada por homologia (Homology-Directed Repair, HDR), inserindo um fragmento de DNA fornecido à célula. No entanto, esta técnica envolve a síntese de uma meganuclease específica de cada fragmento de DNA alvo[12]. Também em 1985, foram descobertas pequenas proteínas que, devido à presença de um ião de zinco que estabiliza a ligação ao DNA e de um conjunto de motivos que se ligam a sequências de três pares de bases específicas do DNA alvo, apresentam uma elevada versatilidade na ligação ao DNA[13], [14]. Em 1996, com a fusão destas proteínas a domínios de clivagem de DNA, foram criadas Zinc Finger Nucleases (ZFN)[15]. Em 2010, novas nucleases programáveis foram concebidas, as nucleases efetoras semelhantes a ativadores de transcrição (Transcription Activator-Like Effector Nucleases, TALEN) [16], enzimas que juntam, também, enzimas de clivagem de DNA a módulos de ligação a nucleótidos de DNA[17], sendo que nesta técnica cada módulo liga-se a um nucleótido, usando para tal apenas dois aminoácidos, o que torna a conceção destas nucleases bastante mais fácil do que as ZFN[18]. Todas estas técnicas envolvem a morosa tarefa de construção de novos domínios ou módulos proteicos para cada sequência alvo a editar[19]. Contudo, um novo sistema viria resolver este problema e revolucionar o mundo da engenharia genética, a técnica CRISPR/Cas.
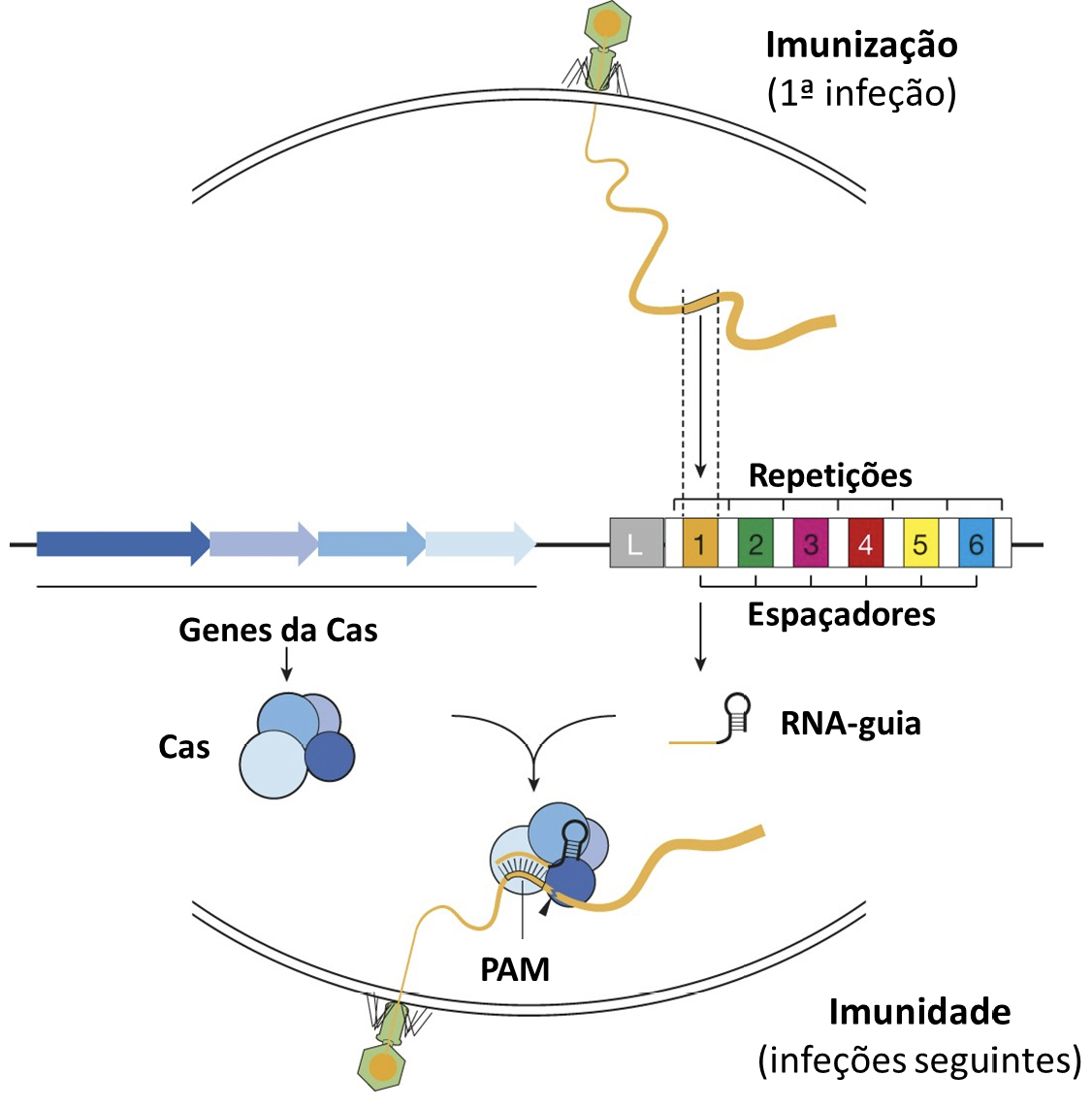
Nos finais da década de 80 do século XX, foram observadas sequências de DNA repetidas no genoma de Escherichia coli, designadas repetições, e espaçadas regularmente por sequências não repetidas derivadas de vírus, os espaçadores[20]. Mais tarde, aquelas repetições foram também identificadas noutros genomas de bactérias e de Archaea (arqueias)[21] e associadas a genes conservados que expressam endonucleases, passando este conjunto de repetições a ser denominado de Repetições Palindrómicas Curtas Agrupadas e Regularmente Interespaçadas (Clustered Regularly Interspaced Short Palindromic Repeats, CRISPR)[22]. Posteriormente, os espaçadores foram, também, encontrados em genomas de fagos e noutros elementos genéticos estranhos[23], [24], [25], levando a que, por fim, o conjunto de repetições CRISPR fosse associado a um sistema de defesa bacteriano contra material genético estranho. Neste sistema, numa primeira infeção por uma dado material genético estranho (fase de imunização), este é fragmentado e inserido entre as repetições, constituindo os tais espaçadores do CRISPR que, numa segunda infeção pelo mesmo material genético (fase de imunidade), irão direcionar a nuclease Cas para o genoma invasor, cortando-o de forma precisa[26]. Para tal, a enzima Cas usa como guia um RNA transcrito dos espaçadores inseridos no seu genoma na fase de imunização, o RNA-guia, que então se liga por complementaridade ao DNA alvo da Cas, o material genético estranho desta segunda infeção[27], [28]. Para que a enzima Cas o reconheça, este DNA alvo tem de estar adjacente a um motivo protoespaçador (Protospacer Adjacent Motif, PAM), fulcral para a clivagem do DNA alvo e para distinguir este DNA dos segmentos anteriormente incluídos no CRISPR[29], [30], [31], [32] (FIGURA 1). Este sistema foi posteriormente adaptado para outras espécies e testado in vitro e in vivo[33], [34], [35], [36], [37], [38], constituindo assim uma tecnologia de edição de genomas bastante mais prática e económica do que as anteriores, pois apenas envolve a síntese de uma pequena molécula de RNA-guia para cada alvo a ser editado, tendo sido utilizado na edição de genomas de praticamente todos os organismos modelo[39].
Relativamente à aplicação da técnica CRISPR/Cas na edição de genomas de espécies vegetais, este sistema tem sido utilizado para melhorar diversas características, como a produtividade e a gestão de stresse biótico e abiótico, em plantas com interesse económico, como o arroz, a cevada, o milho, o trigo, o algodão, o tomateiro, a soja, citrinos, a batateira, a videira, a melancia, a bananeira, a cenoura, leguminosas, a mandioca e a batata-doce[47]. A expressão do gene da nuclease Cas, bem como dos RNA-guia nas células alvo, é suficiente para a modificação do genoma de interesse. Em 2013 iniciaram-se as primeiras edições de genomas de plantas, quer em dicotiledóneas com Arabidopsis thaliana[48], uma planta modelo, quer em monocotiledóneas com o arroz, Oryza sativa[49], [50]. Enquanto que em Arabidopsis a primeira edição genética com esta tecnologia se centrou em provar que também funciona em plantas, tendo os autores mutado com elevada eficiência três genes relacionados com a fenologia da planta[51], no caso do arroz, embora o objetivo da investigação fosse também a prova de conceito, a função dos genes mutados está associada ao controlo de respostas a stresses bióticos e abióticos[52], [53].
Para além do seu interesse económico, o tomateiro é uma espécie ideal para testar técnicas de edição genómica dada a disponibilidade de métodos de transformação eficazes, a caracterização genómica já alcançada nesta espécie e a existência de um grande historial de melhoramento[54]. No que toca ao melhoramento de qualidade, esta planta já foi alvo de várias intervenções usando a técnica CRISPR/Cas, como obtenção de frutos partenocárpicos[55], [56], aumento do tempo de prateleira[57] e aumento de produtividade[58], [59]. Num dos estudos mais impactantes de edição do genoma do tomateiro, foi possível através da técnica CRISPR/Cas mutar a zona autoinibitória da enzima descarboxilase do glutamato[60], responsável pela produção de ácido gama-aminobutírico (Gamma-AminoButyric Acid, GABA) a partir do glutamato, levando à acumulação de GABA no tomate. GABA é um aminoácido não proteico com benefícios para a tensão arterial. Um estudo posterior envolveu a criação de um mutante para quatro genes relacionados com o metabolismo do GABA, levando também à sua acumulação[61]. Graças a estes melhoramentos, o tomate enriquecido em GABA foi o primeiro alimento editado pela técnica CRISPR/Cas a entrar no mercado[62]. No controlo de respostas a stresses, a utilização de dois RNA-guia na edição do gene Mildew Resistant Locus O (MLO), gene com um grande impacto na suscetibilidade ao fungo Oidium neolycopersici, fungo responsável pela doença do oídio no tomateiro, permitiu uma deleção de 48 pb neste gene e, consequentemente, a resistência a este fungo, criando a variedade não transgénica Tomelo[63], [64].
No entanto, a técnica CRISPR/Cas apresenta alguns desafios. Já na fase inicial de experiências de edição genómica por CRISPR/Cas duas abordagens foram testadas relativamente à transformação de plantas com o vetor que contém o gene da Cas e os RNA-guia: em Arabidopsis o método optado foi o floral dip[65], segundo o qual os botões florais nas suas fases iniciais de desenvolvimento são mergulhados numa solução contendo bactérias da espécie Agrobacterium tumefaciens previamente transformadas com o plasmídeo contendo os genes que expressam a endonucleases Cas9 e os RNA-guia utilizados[66]; por outro lado, nos primeiros estudos com o arroz, o método seguido foi a transformação química de protoplastos[67], [68] e o bombardeamento de calli[69] Aqui encontramos um dos desafios da técnica CRISPR/Cas, a escolha do método de transformação da espécie desejada, que terá em conta quer o processo de regeneração, quer a transmissão da mutação induzida às gerações seguintes. Geralmente, as mutações originadas em Arabidopsis por esta técnica, quando o método de transformação utilizado é o floral dip, apresentam uma baixa taxa de transmissão às gerações seguintes, devido à utilização de promotores constitutivos na regulação da expressão da enzima Cas[70]. A adoção de enzimas Cas, cujo gene é regulado por um promotor específico das células germinativas, resolveu este problema[71]. Por outro lado, no caso descrito no arroz a fase de transformação ocorreu em culturas de tecidos utilizando a totipotência das células para potenciar a transmissão da modificação[72]. No entanto, nem todos as plantas de interesse económico são facilmente regeneradas a partir de culturas de tecidos, como as leguminosas, tendo já sido desenvolvidos diferentes protocolos otimizados de regeneração em diferentes espécies[73]. Para espécies recalcitrantes têm sido desenvolvidos novos sistemas vetoriais dos genes necessários ao CRISPR/Cas, como a edição genómica induzida por genes virais, onde vetores baseados em vírus são replicados na planta infetada, propagando a mutação por todo a planta incluindo nas células germinativas, e a edição genómica de linhas elite por cruzamento com linhas mutadas menos recalcitrantes[74]. Relativamente ao complexo Cas-RNA-guia e à sua especificidade, as mutações efetuadas pela enzima Cas em genes inespecíficos constituem um dos grandes problemas da técnica e apoiam alguns argumentos éticos contra a sua utilização, principalmente quando empregue em espécies com genomas grandes e onde a totalidade do genoma não é conhecida[75]. Embora os RNA-guia contenham 20 nucleótidos o mais específicos possível para o gene que se pretende mutar, outros aspetos, como a estrutura do RNA-guia, o local PAM escolhido e a estrutura final do complexo RNA-guia – Cas-gene alvo podem afetar a especificidade do corte efetuado pela endonuclease. A utilização de endonucleases modificadas de elevada fidelidade, rácios Cas/RNA-guia otimizados, plataformas de desenho de RNA-guia mais fiéis e genomas de referência de elevada qualidade podem solucionar este problema. Também a introdução de genes estranhos por HDR após o corte da Cas apresenta grandes dificuldades em eucariotas no que toca à precisão e exatidão da inserção. Algumas das soluções consistem em desligar por mutagénese os mecanismos de NHEJ, ou expressar enzimas de modificação do DNA no genoma de interesse, com o intuito de modificar a sua tipologia e facilitar a HDR, ou ainda a utilização de outros estimulantes deste processo[76].
Em conclusão, embora os organismos modificados pela técnica CRISPR/Cas sejam considerados organismos geneticamente modificados (OGMs) de acordo com a legislação em vigor na União Europeia, o facto é que, ao contrário dos organismos transgénicos em que um gene com função conhecida é transferido de um organismo para o outro, sendo integrado aleatoriamente no genoma do segundo, nos organismos geneticamente editados segmentos muitos específicos do seu DNA são alterados com vista a alterar a função ou expressão do gene alvo, não havendo necessariamente inserção de um gene estranho no genoma do organismo. Aparentemente a técnica CRISPR/Cas apresenta apenas duas grandes preocupações quanto à sua aplicação em biotecnologia vegetal e no melhoramento de plantas: as mutações inespecíficas e a deriva genética. Quanto às primeiras, e como já foi referido, para além desta técnica apresentar uma maior fidelidade relativamente às técnicas de edição genómica anteriores, são atualizadas com frequência novas ferramentas que aumentam a fidelidade da técnica e novos genomas são conhecidos mais profundamente, evitando os erros das mutações inespecíficas. Quanto à deriva genética, embora a introdução de edições no genoma transmissíveis à descendência possa alterar drasticamente o pool genético e causar distúrbios em espécies silvestres, a reversão desta situação com a reintrodução da sequência original do gene editado pode resolver este problema[77]. Assim, embora alguns pormenores e especificidades da técnica CRISPR/Cas possam ser melhoradas, a potencialidade que apresenta para a modificação de genomas será muito importante no melhoramento de plantas num futuro próximo, principalmente com o aumento do número de espécies vegetais com o genoma sequenciado.
[editar] Referências
- ↑ CAPECCHI, M. R., Altering the genome by homologous recombination, Science, 244, 1288-1292. 1989.
- ↑ KHALIL, A. M., The genome editing revolution, Journal of Genetic Engineering and Biotechnology, 18, 1-16. 2020.
- ↑ TUTEJA, N. et al., Recent advances in development of marker-free transgenic plants: regulation and biosafety concern, Journal of Biosciences, 37, 167-197. 2012.
- ↑ CHEN, L., A method for the production and expedient screening of CRISPR/Cas9-mediated non-transgenic mutant plants, Horticulture Research, 5. 2018.
- ↑ KOUL, B., Cisgenics and Transgenics: Strategies for Sustainable Crop Development and Food Security, Springer, p. 107-129. 2022.
- ↑ TRÖDER, S. E. & ZEVNIK, B., History of genome editing: From meganucleases to CRISPR, Laboratory Animals, 56, 60-68. 2022.
- ↑ SHUVALOV, O. et al., Current genome editing tools in gene therapy: new approaches to treat cancer, Current Gene Therapy, 15, 511-529. 2015.
- ↑ TRÖDER, S. E. & ZEVNIK, B., History of genome editing: From meganucleases to CRISPR, Laboratory Animals, 56, 60-68. 2022.
- ↑ ROUET, P. et al., Introduction of double-strand breaks into the genome of mouse cells by expression of a rare-cutting endonuclease, Molecular and Cellular Biology, 14, 8096-8106. 1994.
- ↑ JACQUIER, A. & DUJON, B., An intron-encoded protein is active in a gene conversion process that spreads an intron into a mitochondrial gene, Cell, 41, 383-394. 1985.
- ↑ ROUET, P. et al., Introduction of double-strand breaks into the genome of mouse cells by expression of a rare-cutting endonuclease, Molecular and Cellular Biology, 14, 8096-8106. 1994.
- ↑ TRÖDER, S. E. & ZEVNIK, B., History of genome editing: From meganucleases to CRISPR, Laboratory Animals, 56, 60-68. 2022.
- ↑ KLUG, A., The discovery of zinc fingers and their applications in gene regulation and genome manipulation, Annual Review of Biochemistry, 79, 213-231. 2010.
- ↑ MILLER, J. et al., Repetitive zinc-binding domains in the protein transcription factor IIIA from Xenopus oocytes,, The EMBO Journal, 4, 1609-1614. 1985.
- ↑ KIM, Y-G. et al., Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain, Proceedings of the National Academy of Sciences, 93, 1156-1160. 1996.
- ↑ CHRISTIAN, M. et al., Targeting DNA double-strand breaks with TAL effector nucleases, Genetics, 186, 757-761. 2010.
- ↑ MILLER, J. C., A TALE nuclease architecture for efficient genome editing, Nature Biotechnology, 29, 143-148. 2011.
- ↑ BOGDANOVE, A. J. & VOYTAS, D. F., TAL effectors: customizable proteins for DNA targeting, Science, 333, 1843-1846. 2011.
- ↑ TRÖDER, S. E. & ZEVNIK, B., History of genome editing: From meganucleases to CRISPR, Laboratory Animals, 56, 60-68. 2022.
- ↑ ISHINO, Y. et al., Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product, Journal of Bacteriology, 169, 5429-5433. 1987.
- ↑ MOJICA, F. J. et al., Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria, Molecular Microbiology, 36, 244-246. 2000.
- ↑ JANSEN, R. et al., Identification of genes that are associated with DNA repeats in prokaryotes, Molecular Microbiology, 43, 1565-1575. 2002.
- ↑ BOLOTIN, A. et al., Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin, Microbiology, 151, 2551-2561. 2005.
- ↑ MOJICA, F. J. et al., Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements, Journal of Molecular Evolution, 60, 174-182. 2005.
- ↑ POURCEL, C. et al., CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies, Microbiology, 151, 653-663. 2005.
- ↑ BARRANGOU, R. et al., CRISPR provides acquired resistance against viruses in prokaryotes, Science, 315, 1709-1712. 2007.
- ↑ BROUNS, S. J. et al., Small CRISPR RNAs guide antiviral defense in prokaryotes, Science, 321, 960-964. 2008.
- ↑ MARRAFFINI, L. A. & SONTHEIMER, E. J., CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA, Science, 322, 1843-1845. 2008.
- ↑ DEVEAU, H. et al., Phage response to CRISPR-encoded resistance in Streptococcus thermophilus, Journal of Bacteriology, 190, 1390- 1400. 2008.
- ↑ JINEK, M. et al., A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity, Science, 337, 816-821. 2012.
- ↑ MARRAFFINI, L. A. & SONTHEIMER, E. J., Self versus non-self discrimination during CRISPR RNA-directed immunity, Nature, 463, 568-571. 2010.
- ↑ MOJICA, F. J. et al., Short motif sequences determine the targets of the prokaryotic CRISPR defence system, Microbiology, 155, 733-740. 2009.
- ↑ CONG, L. et al., Multiplex genome engineering using CRISPR/Cas systems, Science, 339, 819-823. 2013.
- ↑ GASIUNAS, G. et al., Cas9–crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria, Proceedings of the National Academy of Sciences, 109, E2579-E2586. 2012.
- ↑ JINEK, M. et al., RNA-programmed genome editing in human cells, Elife, 2, e00471. 2013.
- ↑ MALI, P. et al., RNA-guided human genome engineering via Cas9, Science, 339, 823-826. 2013.
- ↑ WANG, H. et al., One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering, Cell, 153, 910-918. 2013.
- ↑ YANG, H. et al., One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering, Cell, 154, 1370-1379. 2013.
- ↑ TRÖDER, S. E. & ZEVNIK, B., History of genome editing: From meganucleases to CRISPR, Laboratory Animals, 56, 60-68. 2022.
- ↑ BROUNS, S. J. et al., Small CRISPR RNAs guide antiviral defense in prokaryotes, Science, 321, 960-964. 2008.
- ↑ MARRAFFINI, L. A. & SONTHEIMER, E. J., CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA, Science, 322, 1843-1845. 2008.
- ↑ DEVEAU, H. et al., Phage response to CRISPR-encoded resistance in Streptococcus thermophilus, Journal of Bacteriology, 190, 1390- 1400. 2008.
- ↑ JINEK, M. et al., A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity, Science, 337, 816-821. 2012.
- ↑ MARRAFFINI, L. A. & SONTHEIMER, E. J., Self versus non-self discrimination during CRISPR RNA-directed immunity, Nature, 463, 568-571. 2010.
- ↑ MOJICA, F. J. et al., Short motif sequences determine the targets of the prokaryotic CRISPR defence system, Microbiology, 155, 733-740. 2009.
- ↑ MARRAFFINI, L. A., CRISPR-Cas immunity in prokaryotes, Nature, 526, 55-61. 2015.
- ↑ JAGANATHAN, D. et al., CRISPR for crop improvement: an update review, Frontiers in Plant Science, 9, 985. 2018.
- ↑ FENG, Z. et al., Efficient genome editing in plants using a CRISPR/Cas system, Cell Research, 23, 1229-1232. 2013.
- ↑ SHAN, Q. et al., Targeted genome modification of crop plants using a CRISPR-Cas system, Nature Biotechnology, 31, 686-688. 2013.
- ↑ XIE, K. & YANG, Y., RNA-guided genome editing in plants using a CRISPR–Cas system, Molecular Plant, 6, 1975-1983. 2013.
- ↑ FENG, Z. et al., Efficient genome editing in plants using a CRISPR/Cas system, Cell Research, 23, 1229-1232. 2013.
- ↑ SHAN, Q. et al., Targeted genome modification of crop plants using a CRISPR-Cas system, Nature Biotechnology, 31, 686-688. 2013.
- ↑ XIE, K. & YANG, Y., RNA-guided genome editing in plants using a CRISPR–Cas system, Molecular Plant, 6, 1975-1983. 2013.
- ↑ PAN, C. et al., CRISPR/Cas9-mediated efficient and heritable targeted mutagenesis in tomato plants in the first and later generations, Scientific Reports, 6, 1-9. 2016.
- ↑ KLAP, C. et al., Tomato facultative parthenocarpy results from Sl AGAMOUS-LIKE 6 loss of function, Plant Biotechnology Journal, 15, 634-647. 2017.
- ↑ UETA, R. et al., Rapid breeding of parthenocarpic tomato plants using CRISPR/Cas9, Scientific Reports, 7, 1-8. 2017.
- ↑ YU, Q. et al., CRISPR/Cas9-induced targeted mutagenesis and gene replacement to generate long-shelf life tomato lines, Scientific Reports, 7, 1-9. 2017.
- ↑ SOYK, S., & MÜLLER, N. A. et al., Variation in the flowering gene SELF PRUNING 5G promotes day-neutrality and early yield in tomato, Nature Genetics, 49, 162-168. 2017.
- ↑ PIMENTEL, D. & FORTES, A. M., Targeted genome editing using CRISPR-Cas9: applications in fruit quality and stress resilience, Advancement in crop improvement techniques, Woodhead Publishing, Elsevier, p 199-207. 2020.
- ↑ NONAKA, S. et al., Efficient increase of aminobutyric acid (GABA) content in tomato fruits by targeted mutagenesis, Scientific Reports, 7, 1-14. 2017.
- ↑ LI, R. et al., Multiplexed CRISPR/Cas9-mediated metabolic engineering of aminobutyric acid levels in Solanum lycopersicum, Plant Biotechnology Journal, 16, 415-427. 2018.
- ↑ WALTZ, E., GABA-enriched tomato is first CRISPR-edited food to enter market, Nat Biotechnol, 40, 9-11. 2022.
- ↑ PIMENTEL, D. & FORTES, A. M., Targeted genome editing using CRISPR-Cas9: applications in fruit quality and stress resilience, Advancement in crop improvement techniques, Woodhead Publishing, Elsevier, p 199-207. 2020.
- ↑ NEKRASOV, V. et al., Rapid generation of a transgene-free powdery mildew resistant tomato by genome deletion, Scientific Reports, 7, 1-6. 2017.
- ↑ CLOUGH, S. J. & BENT, A. F., Floral dip: a simplified method for Agrobacterium mediated transformation of Arabidopsis thaliana, The Plant Journal, 16, 735-743. 1998.
- ↑ FENG, Z. et al., Efficient genome editing in plants using a CRISPR/Cas system, Cell Research, 23, 1229-1232. 2013.
- ↑ SHAN, Q. et al., Targeted genome modification of crop plants using a CRISPR-Cas system, Nature Biotechnology, 31, 686-688. 2013.
- ↑ XIE, K. & YANG, Y., RNA-guided genome editing in plants using a CRISPR–Cas system, Molecular Plant, 6, 1975-1983. 2013.
- ↑ SHAN, Q. et al., Targeted genome modification of crop plants using a CRISPR-Cas system, Nature Biotechnology, 31, 686-688. 2013.
- ↑ PAUL, J. W. & QI, Y., CRISPR/Cas9 for plant genome editing: accomplishments, problems and prospects, Plant Cell Reports, 35, 1417- 1427. 2016.
- ↑ WANG, Z-P. et al., Egg cell-specific promoter-controlled CRISPR/Cas9 efficiently generates homozygous mutants for multiple target genes in Arabidopsis in a single generation, Genome Biology, 16, 1-12. 2015.
- ↑ PAUL, J. W. & QI, Y., CRISPR/Cas9 for plant genome editing: accomplishments, problems and prospects, Plant Cell Reports, 35, 1417- 1427. 2016.
- ↑ BHOWMIK, P. et al., CRISPR/Cas9 gene editing in legume crops: opportunities and challenges, Legume Science, 3, e96. 2021.
- ↑ SON, S. & PARK, S. R., Challenges facing CRISPR/Cas9-based genome editing in plants, Frontiers in Plant Science, 13, 902413. 2022.
- ↑ ZHANG, F. et al., CRISPR/Cas9 for genome editing: progress, implications and challenges, Human Molecular Genetics, 23, R40-R46. 2014.
- ↑ PAUL, J. W. & QI, Y., CRISPR/Cas9 for plant genome editing: accomplishments, problems and prospects, Plant Cell Reports, 35, 1417- 1427. 2016.
- ↑ ES, I., The application of the CRISPR-Cas9 genome editing machinery in food and agricultural science: current status, future perspectives, and associated challenges, Biotechnology Advances, 37, 410-421. 2019.
Criada em 29 de Novembro de 2022
Revista em 16 de Fevereiro de 2023
Aceite pelo editor em 15 de Março de 2023

