Proteínas Virais no Protein Data Bank
Referência : Pinto, A. V., Oliveira, A., Jerves, C., Mendonça, F. L., Coutinho, I. F. M., Coimbra, J. T. S., Sousa, J.P.M., Cabrera-Trujillo, J. J., Teixeira, L., Virgas, M. F., Ferreira, P., Paiva, P., Fonseca, R., Neves, Rui P. P., Magalhães, A. L., Fernandes, P.A., Ramos, M.J., (2020) Proteínas Virais no Protein Data Bank, Rev. Ciência Elem., V8(2):022
Autores: Alexandre V. Pinto, Ana N. L. Oliveira, Carola Jerves, Filipa Lopes de Mendonça, Inês F. M. Coutinho, João T. S. Coimbra, João Sousa, Jorge J. Cabrera-Trujillo, Luís Teixeira, Matilde F. Viegas, Pedro Ferreira, Pedro Paiva, Rita Fonseca, Rui P. P. Neves, Alexandre L. Magalhães, Pedro Alexandrino Fernandes e Maria João Ramos
Editor: José Ferreira Gomes
DOI: [http://doi.org/10.24927/rce2020.022]

Descoberto um novo vírus, o SARS-CoV-2, em quatro meses foi já possível determinar 182 estruturas de um número muito reduzido de proteínas deste vírus, isoladas ou ligadas a outras entidades, com diferentes técnicas e resoluções. Apresentam-se visualizações adaptadas de algumas proteínas com estruturas já depositadas no Banco de Dados de Proteínas (Protein Data Bank) por cientistas de todo o mundo e através do qual todos colaboram.
Estruturas 3D de proteínas
As proteínas são macromoléculas constituídas fundamentalmente por aminoácidos, que fazem igualmente parte de todos os sistemas biológicos como sejam os vírus, as bactérias, as leveduras, as plantas, as moscas e os animais. O nome “proteína” vem da palavra grega proteos que significa “primário” ou “primeiro lugar”. Nas proteínas são reconhecidos 4 níveis de estruturas esquematizados na FIGURA 1.
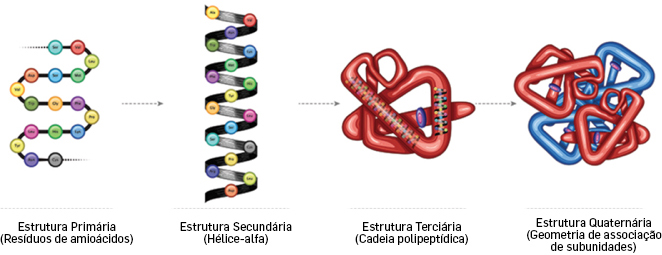
O nível mais simples da estrutura proteica, a estrutura primária, é simplesmente a sequência de aminoácidos de uma cadeia de polipéptidos. Segue-se a estrutura secundária que se refere a motivos repetitivos dentro da proteína tais como hélices ou folhas, que se formam devido a pontes de hidrogénio formadas entre os átomos da cadeia principal da proteína. A estrutura terciária (muitas vezes referida como tridimensional ou 3D) reflete o enrolamento da cadeia de aminoácidos ou, muito simplesmente, a sua conformação global. Finalmente, a estrutura quaternária das proteínas refere-se ao modo como as várias cadeias proteicas (quando existentes) se dispõem entre si.
O Banco de Dados de Proteínas – Protein Data Bank (PDB)
O Protein Data Bank (PDB)[1] é um repositório de informação estrutural 3D de macromoléculas biológicas, nas quais se inserem as proteínas. Estas moléculas para além de fazerem parte da vida de todos os organismos como sejam as bactérias, plantas e animais, constituem também uma parte fundamental dos vírus.
O PDB foi estabelecido em 1971 no Brookhaven National Laboratory, EUA, contendo, originalmente, 7 estruturas. Neste momento, contém 164 174 estruturas macromoleculares biológicas e este número aumenta diariamente. O Research Collaboratory for Structural Bioinformatics (RCSB) tornou-se responsável pela gestão do PDB em 1998. Posteriormente, em 2003, foi criada a wwPDB ou seja a organização que supervisiona atualmente o PDB, tendo por função a manutenção de um único arquivo de dados estruturais macromoleculares do PDB, livre e publicamente disponível para a comunidade global. A informação estrutural que figura no PDB é normalmente obtida por biólogos e bioquímicos de todo o mundo, maioritariamente através de experiências de cristalografia de raios X, espetroscopia de ressonância magnética nuclear (RMN), crio-microscopia eletrónica (crio-EM), difração de neutrões ou mesmo modelação molecular. Os autores destas experiências depositam os seus resultados no arquivo do PDB sendo o seu conteúdo acessível livremente na internet através do site www.rcsb.org. As estruturas que figuram no PDB vão desde proteínas e ácidos nucleicos até máquinas moleculares complexas como o ribossoma. É importante compreender que a função de uma proteína está diretamente ligada à sua estrutura 3D e que o seu estudo tem um papel fundamental na saúde humana, na doença e na descoberta de fármacos.
As proteínas do vírus da COVID-19 no PDB>
SARS-CoV-2 é o vírus causador da pandemia COVID-19[2]. Os vírus, em geral, e este, em particular, são estruturas simples quando comparados com todos os organismos vivos. Assim, o vírus SARS-CoV-2 é constituído por uma membrana viral, uma cadeia simples de RNA e quatro proteínas estruturais denominadas proteína E (do inglês Envelope), proteína M (do inglês Membrane), proteína N (do inglês Nucleocapside) e proteína S (do inglês Spike), para além de várias outras proteínas codificadas no RNA. A proteína N protege o material genético e as três proteínas, S, M e E, estruturam o envelope viral[3]. Apesar de só muito recentemente se terem iniciado estudos de estruturas 3D das proteínas do SARS- -CoV-2, o PDB contém já 182 entradas obtidas nos últimos 4 meses que, de uma forma ou de outra, se relacionam com aquele número muito reduzido de proteínas, isoladas ou ligadas a outras entidades, com diferentes técnicas e resoluções.
Arte na Ciência com tema nas proteínas do vírus da COVID-19
A ciência e a arte são frequentemente consideradas como disciplinas distintas, sendo que, de um modo geral, se considera que uma pessoa não pode interessar-se seriamente por ambas a não ser que o interesse por uma esteja relacionado, de alguma forma, com o trabalho na outra. No entanto, na realidade, os cientistas participam e produzem arte a todos os níveis e em todos os meios. De acordo, com esta visão do mundo, já Albert Einstein afirmou que “os grandes cientistas são também grandes artistas”. Assim, e de acordo com as palavras deste grande mestre, tentamos encontrar dentro de nós os artistas que poderiam relacionar as proteínas do vírus que provoca a tão indesejada COVID-19, com algo harmonioso e visualmente agradável que, simultaneamente, dê a conhecer um pouco da maquinaria microscópica que transtorna a vida. Com as imagens que se seguem, conseguimos vislumbrar pequenos detalhes, pequenas imagens, pequenas composições desta sinfonia e, através delas, imaginar a inimaginável simplicidade sobre a qual o SARS-CoV-2 está construído.
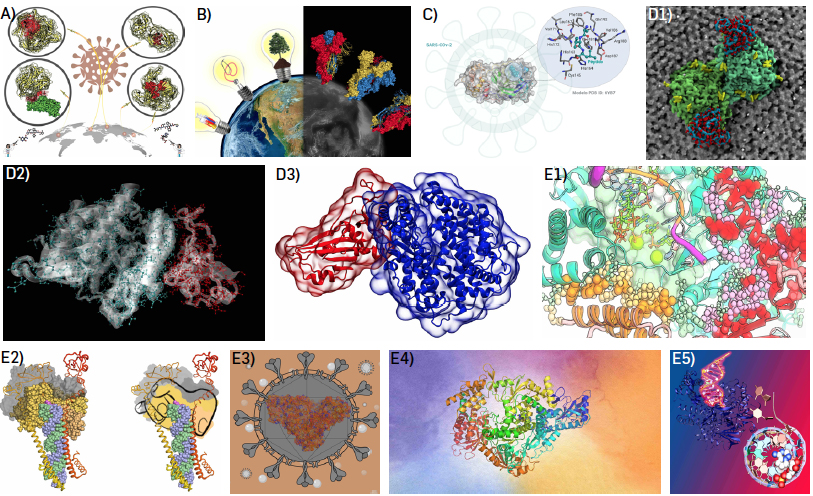
As coordenadas 3D retiradas do PDB das estruturas proteicas deste vírus fornecem- -nos apenas as geometrias que posteriormente trabalhamos para criarmos visualizações não só apelativas como também informativas das funções que as estruturas das proteínas comandam. Na realidade, as estruturas das proteínas estabelecem as bases das suas interações com outras moléculas do organismo e, por conseguinte, determinam as suas funções. Baseadas neste facto tão importante, as imagens aqui apresentadas tentam dar a conhecer ao leitor a maquinaria atómica do vírus através das suas proteínas, informar sobre as suas funções e transmitir uma consciência remota da dimensão do que sobre elas ignoramos e que, provavelmente, nunca chegaremos a compreender completamente.

No entanto, e para além das suas funções artísticas já aqui documentadas, estas visualizações do mundo microscópico podem ser essenciais para a investigação de quem trabalha com estes sistemas biológicos. Como exemplo, examine-se a imagem C da FIGURA 2 deste texto. Ela representa a protease principal do vírus SARS-CoV-2 (MPro), responsável pela clivagem das poliproteínas do mesmo. Durante este processo, são libertadas proteínas virais funcionais e enzimas-chave para a replicação do vírus. Assim, o bloqueio da MPro impedirá a continuidade do SARS-CoV-2. E, nesse sentido, esta mesma imagem está a ser utilizada para estabelecer o mecanismo catalítico da MPro, tendo como base o centro ativo da proteína que se encontra em destaque à direita da imagem e que engloba todos os resíduos de aminoácidos importantes a considerar. Outras imagens têm uma finalidade pedagógica, embora não investigativa, ou ainda política, como seja a imagem A da FIGURA 2 que tenta chamar a atenção para as representações 3D de quatro proteínas, alvos promissores de fármacos contra o vírus SARS-CoV-2 causador da COVID-19. Na fila de cima, à esquerda, está a RNA polimerase dependente de RNA, que é indispensável para a replicação do vírus in vivo; na fila de cima, à direita, temos a protease principal (MPro) do SARS-CoV-2, já considerada no exemplo anterior; na fila de baixo, à esquerda, está representada a glicoproteína spike (S, espícula) fundamental para a entrada do vírus nas células do hospedeiro; e representada a verde temos a enzima conversora da angiotensina 2 (hACE2) que é o recetor à qual a glicoproteína S se liga nas células humanas; por último, na fila de baixo, à direita, está representada a protease do tipo papaína (PLpro) que, tal como a MPro, tem como função processar poliproteínas em proteínas funcionais, ajudando também o vírus a evadir-se da resposta imune do hospedeiro. Ainda na mesma imagem vemos também uma cientista, que após semanas de estudo científico, dispara moléculas com potencial terapêutico com uma arma cronicamente subfinanciada.
Referências
- ↑ BERMAN, H.M. et al. The Protein Data Bank, Nucleic Acids Research, 28, 235-242, 2000.
- ↑ ANDERSEN, K.G. et al. The proximal origin of SARS-CoV-2, Nature Medicine, 26, 450–452, 2020.
- ↑ LI, G. et al. Coronavirus infections and immune responses, J. Med. Virology, 92, 424-432, 2020.
Recursos relacionados disponíveis na Casa das Ciências:
Criada em 19 de Maio de 2020
Revista em 20 de Maio de 2020
Aceite pelo editor em 22 de Junho de 2020
