Enzima TMPRSS2
Referência : Santos, F. L., Fernandes, P. A., Ramos, M. J., (2021) Enzima TMPRSS2, Rev. Ciência Elem., V9(4):065
Autor: Filipa Leça Santos, Pedro A. Fernandes e Maria João Ramos
Editor: João Nuno Tavares
DOI: [https://doi.org/10.24927/rce2021.065]

Resumo
A enzima protease serina transmembranar 2 (TMPRSS2) é um elemento necessário à infeção pelo vírus SARS-CoV-2, o vírus causativo da pandemia COVID-19. A TMPRSS2 é, portanto, um alvo importante para novos fármacos que podem tratar a infeção por SARS-CoV-2.
Após a Organização Mundial de Saúde declarar a COVID-19 uma pandemia, a 11 de março de 2020, a vida como se conhece sofreu muitas alterações. Até hoje, a humanidade sofreu muitas perdas, tendo-se registado, em julho de 2021, cerca de 172 milhões de casos de infeção por SARS-CoV-2, e destes, 3 milhões não sobreviveram, tornando-se esta pandemia uma ameaça mundialmente significativa[1]. Como em qualquer pandemia, a melhor arma para a combater é a Ciência. Através desta, surgiram as vacinas, que previnem, em média, quatro a cinco milhões de mortes por ano[2].
É crucial ter em mente que um vírus, como o SARS-CoV-2, sofre rápidas mutações. Isto significa que cada vez que o vírus se instala num novo hospedeiro tem uma probabilidade de sofrer uma mutação, que pode ou não ser mais patológica ou mais infeciosa. Os vírus mutantes mais contagiosos tornam-se dominantes na população dando origem a novas estirpes virais. Em apenas um ano de pandemia, já se desenvolveram a estirpe inglesa, a brasileira, a sul-africana e a indiana. Constata-se pois, que este vírus é propício a mutações e que apesar do muito esforço e dedicação por parte de todos os investigadores, algumas dessas alterações genómicas acabam por beneficiar o vírus e, consequentemente, prolongar a sua permanência na população, com o risco de fazer com que todas as vacinas já criadas e aprovadas possam vir a ser ineficientes, ou menos eficientes, contra as novas variantes. Assim sendo, é muito importante controlar a propagação do vírus para que a probabilidade de mutações seja o mais diminuta possível. Visto isto, quanto mais conhecimento e estratégias forem adquiridas maior será a vantagem do ser humano no combate à pandemia atual.
Muitos foram os temas já abordados e investigados. No entanto, ainda há vários caminhos por explorar; um deles é o estudo da enzima protease serina transmembranar 2, mais conhecida por TMPRSS2. Apesar de esta enzima não pertencer ao SARS-CoV-2, foi constatado que ela tem um papel fundamental na infeção do hospedeiro, e, por esse motivo, poderá ser uma potencial via ao combate da pandemia atual.
Enzima TMPRSS2
As proteases são enzimas que clivam ligações peptídicas, que são as ligações formadas entre os aminoácidos que constituem as proteínas[3]. O processo é chamado clivagem proteolítica, um mecanismo comum de ativação ou inativação de enzimas[4].
As proteases serina são endopeptidases — clivam as partes internas das proteínas- e apresentam um resíduo de serina no centro ativo que atua como nucleófilo. As proteases serinas são caracterizadas por um centro ativo que contém três aminoácidos altamente conservados - serina (Ser), histidina (His) e aspartato (Asp) - formando a chamada tríade catalítica[5]. Com base na sua preferência para substratos, as proteases serina subdividem- -se em vários tipos, tais como tipo tripsina ou quimotripsina, entre outros[6]. A protease serina transmembranar 2 (TMPRSS2) é uma protease serina transmembranar do tipo II[7], codificada pelo gene TMPRSS2. Esta enzima pertence ao tipo tripsina e, estas enzimas, normalmente, clivam ligações peptídicas que envolvem aminoácidos de lisina ou arginina4.
Estrutura da enzima TMPRSS2
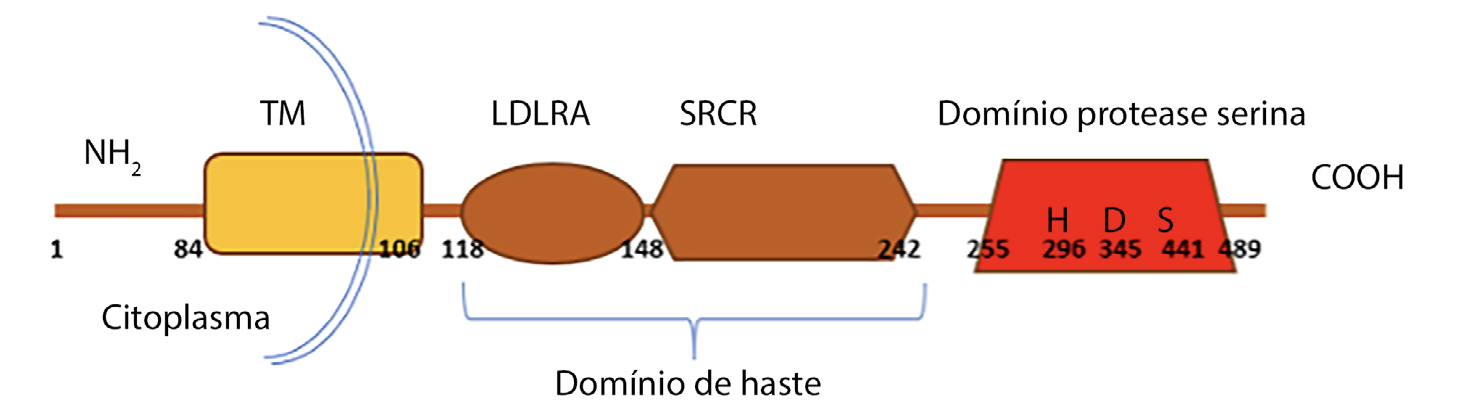
De acordo com o Universal Protein Resource “UniProtKB” database[8], a enzima TMPRSS2 é constituída por 492 aminoácidos. Contém um domínio transmembranar do tipo II; um domínio recetor LDL (do inglês low-density lipoprotein) classe A (LDLRA), aminoácidos 112-149, que contém um centro de coordenação para um ião Ca2+; um domínio recetor rico em cisteína (SRCR), aminoácidos 150-242, que está envolvido na ligação a outras moléculas na superfície celular ou no meio extracelular; um domínio de protease serina, aminoácidos 256-489, e um local de clivagem, aminoácidos 255-256[9], [10], que o domínio protease serina tem de (auto)clivar para que a enzima se torne ativa. O domínio protease serina possui uma tríade catalítica essencial para a atividade proteolítica constituída pelos aminoácidos serina441, histidina296 e aspartato345, localizados numa cavidade que liga o substrato e denominada de centro ativo da enzima[11].
A região N-terminal da enzima encontra-se localizada no citoplasma e, junto a este, situa- se o domínio transmembranar hidrofóbico que pode interagir com componentes citoesqueléticos e moléculas de sinalização, podendo ser importante para o correto transporte intracelular do péptido[12]. Já os domínios LDLRA, SRCR e o domínio protease serina localizam- se no espaço extracelular. Os domínios LDLRA e SRCR constituem um domínio denominado de haste, que pode participar em interações proteína-proteína[13]. O domínio catalítico cliva recetores de membrana celular, fatores de crescimento, citoquinas e componentes da matriz extracelular[14]. Este domínio sofre autoclivagem, secreção no epitélio e interage com as proteínas da superfície celular, a matriz extracelular e as proteínas das células vizinhas[15]. Na FIGURA 1, pode-se observar um esquema ilustrativo da proteína TMPRSS2.
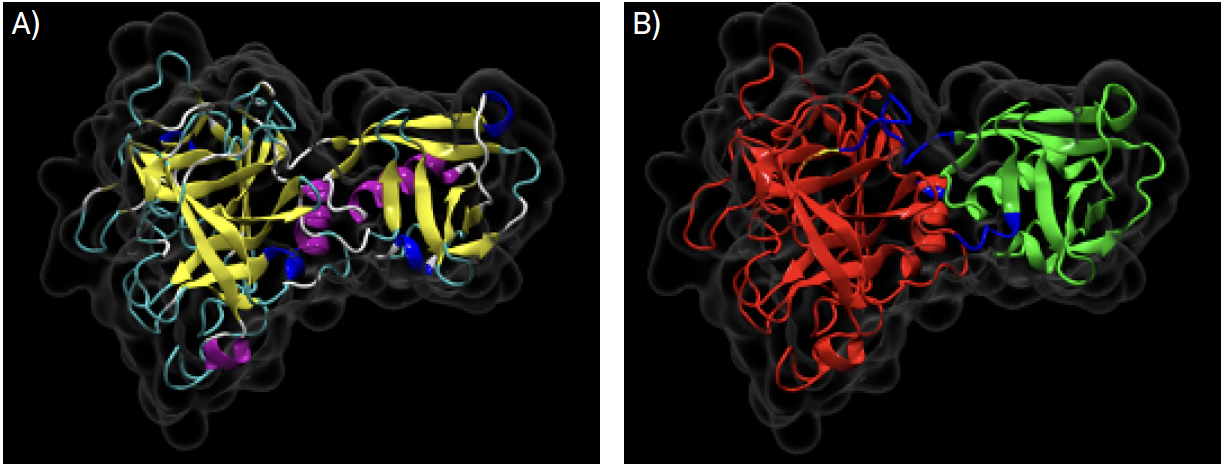
Em termos de estrutura tridimensional, apenas a estrutura do domínio rico em cisteínas e domínio protease serina foram determinados na estrutura depositada na base de dados Protein Data Bank, com o código 7MEQ[16]. A estrutura terciária destes domínios é constituída por hélices-α, folhas-β, hélices 3.10, pontes-β, ganchos (em inglês turns) e enrolamentos desordenados (em inglês random coils) (FIGURA 2A)). Na FIGURA 2B), pode-se observar a representação de tais domínios.
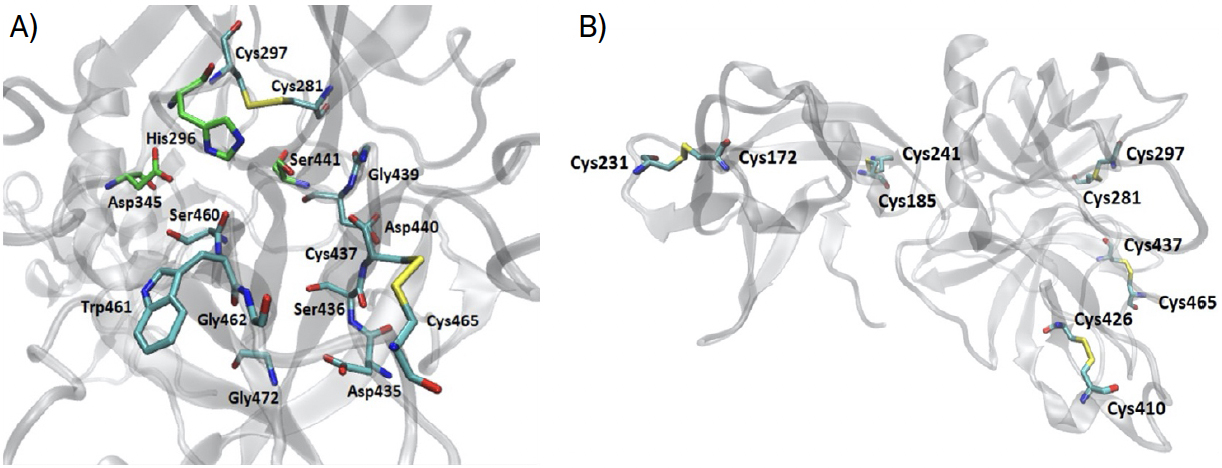
A tríade catalítica (His296, Asp345 e Ser441) localiza-se no centro ativo com a Ser441 de um lado e a His296 e o Asp345 do outro lado da cavidade onde se liga o substrato, sendo esta importante no encaixe do aminoácido a clivar[17]. Os resíduos Asp435, Gly462 e Gly472 criam nesta cavidade um local carregado negativamente e a combinação de Ser441, Gly462 e Gly472 formam uma cavidade hidrofóbica profunda para acomodar aminoácidos hidrofóbicos do substrato5. A cavidade oxianiónica característica das enzimas proteases serina é formada por Gly439 e Ser441. O local de ligação do substrato é formado pelos resíduos Ser460, Trp461 e Gly462, que se espera que formem uma folha antiparalela com a estrutura de base dos resíduos P1-P3 dos seus substratos[18].. Na FIGURA 3A), é evidenciado o centro ativo da protease serina e os aminoácidos que formam a cavidade onde se liga a região clivável do substrato. Esta estrutura é estabilizada por cinco pontes dissulfureto, onde as pontes formadas por Cys172–Cys231 e Cys185–Cys241 encontram-se no domínio SRCR e as pontes formadas por Cys281–Cys297, Cys410–Cys426 e Cys437–Cys465 encontram-se no domínio protease serina[19]., FIGURA 3B).
Função
Em termos de função, a enzima TMPRSS2 tem sido associada a processos fisiológicos e patológicos tais como digestão, remodelação de tecidos, coagulação do sangue, fertilidade, respostas inflamatórias, invasão de células tumorais, apoptose e dor[20]. No entanto, a sua função específica permanece ainda desconhecida[21].
Papel da enzima TMPRSS2 na infeção viral pelo SARS-CoV-2
Para que o vírus SARS-CoV-2 infete uma célula, a proteína de espícula (do inglês spike) necessita de se ligar a um recetor da célula hospedeira humana denominado ACE2 (do inglês Angiotensin Converting Enzyme 2)[22]. Após a ligação entre ambas, há a necessidade de ocorrer clivagem da proteína de espícula, por parte da enzima TMPRSS2, para que a fusão entre as membranas celulares viral e hospedeira tenha lugar (FIGURA 4). A clivagem da proteína de espícula expõe uma região denominada “péptido de fusão”, que permite a entrada viral. Este processo envolve uma mudança estrutural na proteína de espícula. Após a entrada, o genoma viral, que tem a forma de RNAmensageiro, é libertado no citoplasma celular do hospedeiro. Estas alterações conformacionais e o processo de fusão requerem a ação de proteases celulares, cuja disponibilidade é uma etapa limitadora de velocidade na entrada do coronavírus[23]. Em particular, as proteases serina transmembranares do tipo II são ancoradas nas membranas citoplasmáticas e a enzima TMPRSS2 é uma delas. A protease pulmonar TMPRSS2 cliva a proteína de espícula em múltiplos locais, para que a fusão entre as membranas celulares viral e hospedeira possa ocorrer[24]. Este evento provoca a diminuição da sensibilidade viral à inibição através da neutralização de anticorpos, ou seja, conferindo resistência ao processo[25].
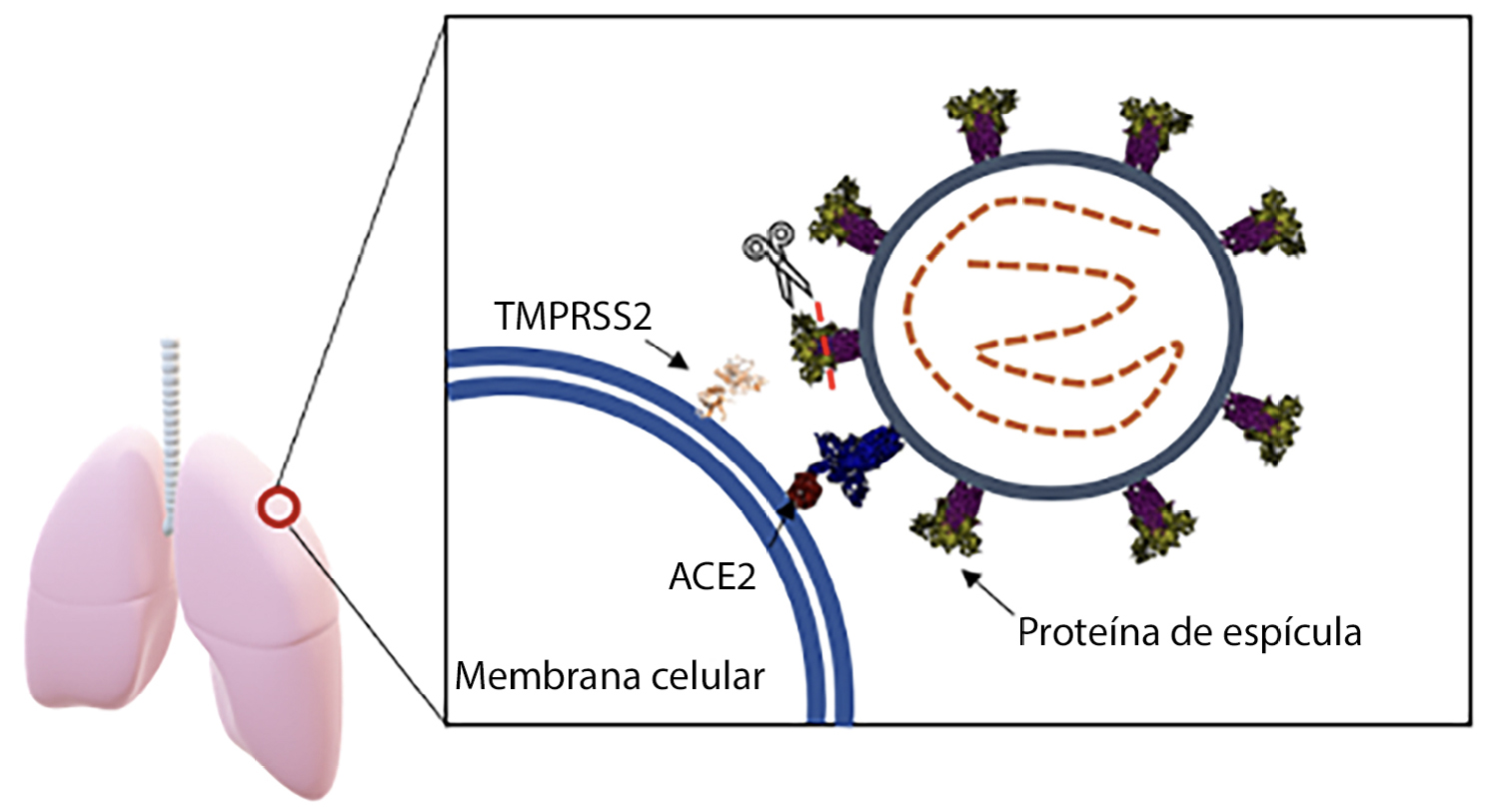
Como a enzima TMPRSS2 subsiste dentro e fora do pulmão, pode contribuir para a propagação extrapulmonar do vírus, o que justifica uma maior exploração desta enzima como um potencial alvo para limitar a propagação viral e, consequentemente, a infecciosidade. De facto, já foram realizados testes in vitro e in silico que demonstraram que a inibição da TMPRSS2 diminui a infeção da célula hospedeira, e por isso, a comunidade científica tem insistido na procura e desenvolvimento de inibidores para esta enzima para que estes possam ser administrados em humanos[26], [27]. Assim, será possível combater a infeção pelo vírus SARS-CoV-2 de forma eficaz.
Referências
- ↑ https://www.worldometers.info/coronavirus/.
- ↑ https://www.who.int/news-room/facts-in-pictures/detail/immunization.
- ↑ HEDSTROM, L., Serine protease mechanism and specificity, Chemical reviews, 102, 12, 4501-4524. 2002.
- ↑ HEDSTROM, L., Serine protease mechanism and specificity, Chemical reviews, 102, 12, 4501-4524. 2002.
- ↑ SINGH, N. et al., Structure-based drug repositioning over the human TMPRSS2 protease domain: search for chemical probes able to repress SARS-CoV-2 Spike protein cleavages, European Journal of Pharmaceutical Sciences, 153, 105495. 2020.
- ↑ SINGH, N. et al., Structure-based drug repositioning over the human TMPRSS2 protease domain: search for chemical probes able to repress SARS-CoV-2 Spike protein cleavages, European Journal of Pharmaceutical Sciences, 153, 105495. 2020.
- ↑ KISHK, S. M. et al., Molecular Insights into Human Transmembrane Protease Serine-2 (TMPS2) Inhibitors against SARS-CoV2: Homology Modelling, Molecular Dynamics, and Docking Studies, Molecules, 25, 21. 2020.
- ↑ CHIKHALE, R.V. et al., Identification of potential anti-TMPRSS2 natural products through homology modelling, virtual screening and molecular dynamics simulation studies, J Biomol Struct Dyn, 1-16. 2020.
- ↑ KISHK, S. M. et al., Molecular Insights into Human Transmembrane Protease Serine-2 (TMPS2) Inhibitors against SARS-CoV2: Homology Modelling, Molecular Dynamics, and Docking Studies, Molecules, 25, 21. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ FRASER, B. et al., Structural Genomics Consortium (SGC), Crystal structure of human TMPRSS2 in complex with Nafamostat. 2021.
- ↑ KISHK, S. M. et al., Molecular Insights into Human Transmembrane Protease Serine-2 (TMPS2) Inhibitors against SARS-CoV2: Homology Modelling, Molecular Dynamics, and Docking Studies, Molecules, 25, 21. 2020.
- ↑ KISHK, S. M. et al., Molecular Insights into Human Transmembrane Protease Serine-2 (TMPS2) Inhibitors against SARS-CoV2: Homology Modelling, Molecular Dynamics, and Docking Studies, Molecules, 25, 21. 2020.
- ↑ KISHK, S. M. et al., Molecular Insights into Human Transmembrane Protease Serine-2 (TMPS2) Inhibitors against SARS-CoV2: Homology Modelling, Molecular Dynamics, and Docking Studies, Molecules, 25, 21. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ HOFFMANN, M. et al., SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor, cell, 181, 2, 271-280. e8. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ THUNDERS, M. & B. DELAHUNT, Gene of the month: TMPRSS2 (transmembrane serine protease 2), Journal of clinical pathology, 73, 12, 773-776. 2020.
- ↑ GLOWACKA, I. et al., Evidence that TMPRSS2 activates the severe acute respiratory syndrome coronavirus spike protein for membrane fusion and reduces viral control by the humoral immune response, J Virol, 85, 9, 4122-34. 2011.
- ↑ GLOWACKA, I. et al., Evidence that TMPRSS2 activates the severe acute respiratory syndrome coronavirus spike protein for membrane fusion and reduces viral control by the humoral immune response, J Virol, 85, 9, 4122-34. 2011.
- ↑ HU, X. et al., Discovery of TMPRSS2 inhibitors from virtual screening, bioRxiv, 2020.12.28.424413. 2020.
- ↑ PASZTI-GERE, E. et al., In vitro characterization of TMPRSS2 inhibition in IPEC-J2 cells, Journal of Enzyme Inhibition and Medicinal Chemistry, 123-129. 2016.
Criada em 5 de Agosto de 2021
Revista em 23 de Agosto de 2021
Aceite pelo editor em 15 de Dezembro de 2021
