Coronavírus
Referência : Esteves, P.J., (2020) Coronavírus, Rev. Ciência Elem., V8(3):038
Autor: Pedro José Esteves
Editor: José Ferreira Gomes
DOI: [http://doi.org/10.24927/rce2020.038]

Resumo
Os coronavírus podem originar infeções muito graves ou letais nas populações humanas. Em 2002, surgiu um coronavírus, o SARS-CoV que terá tido a sua origem em Morcegos na China, sendo transmitido a um mamífero, a Civeta, que depois infetou os humanos e originou um surto de uma infeção respiratória aguda grave, o SARS. Em 2012, surgiu uma nova doença, a síndrome respiratória do Médio Oriente designada por MERS, provocada pelo coronavírus MERS-CoV que terá tido a sua origem também em morcegos, que o terão transmitido a Camelos que finalmente o transmitiram aos humanos. Em 2019, apareceu um terceiro coronavírus, designado por SARS-CoV2, que provoca um síndrome respiratório agudo designado por COVID-19. O SARS-CoV2 terá também tido a sua origem em morcegos na China e pensa-se que o Pangolim será a espécie intermediária. Atualmente, a hipótese mais provável é que o SARS-CoV2 seja resultado de uma recombinação natural. A recombinação ocorre quando uma célula de um hospedeiro é infetada por várias estirpes de um vírus, que trocam informação genética entre si e originam um vírus recombinante. De facto, ao longo do seu genoma, o SARS-CoV2 apresenta uma maior similaridade com a estirpe detetada no morcego-ferradura, mas no domínio de ligação ao recetor humano a semelhança é muito maior com a estirpe detetada no Pangolim. Uma caracteristica única do SARS-CoV2 é a inserção de quatro aminoácidos na proteína da espícula (Spike) que vai criar um sítio de clivagem para uma enzima, a furina. Algumas proteínas estão inativas quando são sintetizadas, e só se tornam ativas quando partes da proteína são clivadas pela furina, como será o caso do SARS-CoV2. Existem várias evidências de que o SARS-CoV2 consegue replicar e transmitir-se em vários mamíferos, o que sugere que o vírus possa continuar a circular em reservatórios animais. A probabilidade de ocorrerem novas pandemias com um coronavírus, ou qualquer outro vírus, é muito elevada. Algumas das medidas para diminuir essa probabilidade são a não destruição das florestas virgens ou pouco exploradas, o conhecimento dos vírus que circulam na natureza e o impedimento da introdução de espécies exóticas que trazem consigo novos agentes patogénicos.
As infeções de coronavírus no Homem
O coronavírus pertence à subfamília Coronavirinae, família Coronaviridae. São vírus de RNA de cadeia simples, de polaridade positiva e o seu genoma encontra-se dentro duma cápside rodeada exteriormente por um invólucro essencialmente lipídico. Este invólucro possui quatro proteínas estruturais das quais se destaca a proteína da espícula (spike). A proteína da espícula forma protuberâncias para o exterior do vírus, o que lhe confere o aspeto típico de coroa e do qual deriva o nome Coronavírus (do Latim, corona). O primeiro coronavírus foi descoberto na década de 1930 na galinha[1]. Passaram trinta anos até que, nos anos de 1960, foi identificado pela primeira vez um coronavírus em humanos[2]. Desde aí, e até ao momento, identificaram-se no Homem sete coronavírus que provocam sintomas. Quatro deles são endémicos no homem (designados 229E, NL63, OC43 e HKU1), ou seja, circulam nas populações humanas causando infeções ligeiras[3]. Até 2002, as infeções em humanos provocadas por este tipo de vírus não eram muito graves e por isso não eram uma das prioridades das autoridades de saúde. Em 2002, surgiu um coronavírus, na província chinesa de Guangdong, que originou um surto de uma infeção respiratória aguda grave, o que levou à designação de SARS (Severe Acute Respiratory Syndrome)-CoV4. Este surto matou 774 pessoas em todo o mundo antes de ser controlado4. Uma década depois, em 2012, surgiu uma nova doença, a síndrome respiratória do médio oriente, provocada por um outro coronavírus, designado por MERS (Middle East Respiratory Syndrome)-CoV, que foi identificado pela primeira vez na Arábia Saudita[4]. O MERS-CoV já foi detectado em 27 países, matou até ao momento 858 pessoas e ainda continua activo (o último caso mortal ocorreu em Fevereiro deste ano). Como diz um velho ditado português não há duas sem três, e assim, no fim de 2019, apareceu um novo vírus designado por SARS-CoV2 ou HCoV- 19, que provoca um síndrome respiratório agudo designado por COVID-19[5] e que já infetou mais de 6 milhões de pessoas e matou em todo o mundo mais de 360000 pessoas, tendo ganho o estatuto de pandemia de acordo com a Organização Mundial de Saúde. A principal razão para que o SARS-CoV2 se tenha tornado uma pandemia ao contrário do SARS-CoV e do MERS-CoV é a diferença na taxa de mortalidade. O SARS-CoV2 provoca muito menos mortalidade e uma percentagem muito elevada de infetados é assintomática, o que permite uma muito maior transmissão e dispersão do vírus.
A passagem dos vírus dos morcegos para o homem
Os três coronavírus que provocaram mortalidades em humanos, SARS-CoV, MERS-CoV e SARS- CoV2, apresentam uma grande similaridade com coronavírus detetados em morcegos, o que sugere que estes vírus tenham tido origem em morcegos[6]. De igual modo, todos eles terão usado outros mamíferos como hospedeiros intermediários antes de infetar o homem (FIGURA 1). O SARS-CoV terá tido a sua origem em morcegos de uma caverna na província chinesa de Yunnan, sendo depois transmitido a um pequeno mamífero asiático, a Civeta, que é muito apreciada na China onde é mantido em jaulas para alimentação humana. Curiosamente, este animal é também mantido em cativeiro porque as suas fezes são usadas para produzir café gourmet em vários países asiáticos. Essas Civetas terão sido vendidas nos mercados chineses de Guandong, tendo sido o vírus transmitido a humanos[7]. O MERS-CoV terá tido a sua origem em morcegos que os terão transmitidos a camelos. A maioria das infeções de MERS terão ocorrido entre humanos, existindo, no entanto, várias evidências de que o Camelo é um reservatório do MERS-CoV e, por isso, uma das fontes de infeção da MERS nos humanos, tornando difícil a completa eliminação da doença[8]. O SARS-CoV2 também terá tido a sua origem em morcegos e embora o percurso que terá levado este vírus ao homem não seja ainda perfeitamente conhecido, existem algumas suspeitas. A deteção de coronavírus muito semelhantes ao SARS-CoV2 no Pangolim sugere que terá sido esta espécie o intermediário[9]. No entanto, os vírus não são exatamente iguais e só depois da pesquisa de coronavírus em outros animais é que vamos ter a certeza do animal intermediário. O primeiro surto do SARS-CoV2 terá ocorrido no mercado de Wuhan (província de Hubei) na China, onde vários animais selvagens são mantidos e abatidos para consumo humano. O Pangolim é vendido regularmente para fins medicinais e é apreciado na gastronomia chinesa.
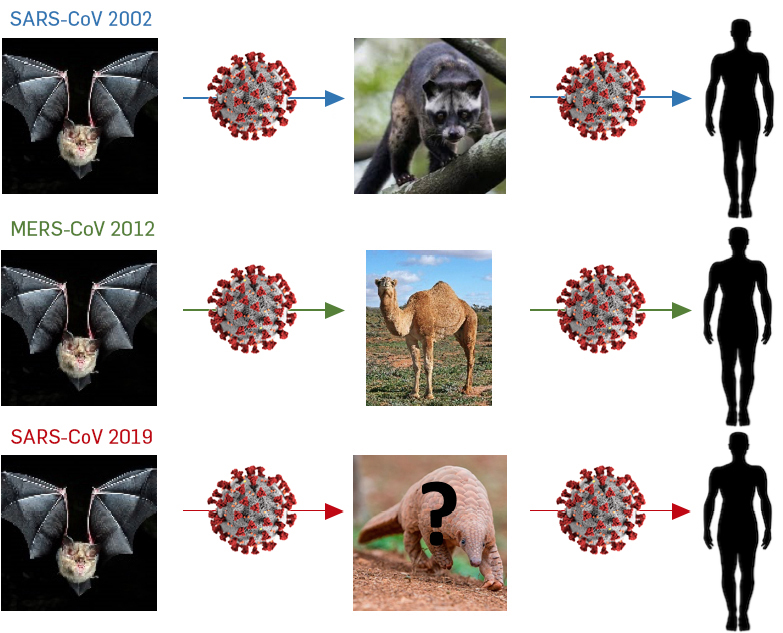
A recente demonstração de que o SARS-CoV2 consegue replicar muito bem em furões e gatos, que os gatos transmitem o vírus entre si, e a recente identificação do SARS-CoV2 em tigres no zoo de Nova Iorque, que terá sido transmitido pelo tratador, sugere que, tal como no MERS-CoV, o vírus possa continuar a circular em reservatórios animais contribuindo para a manutenção do vírus em circulação[10].
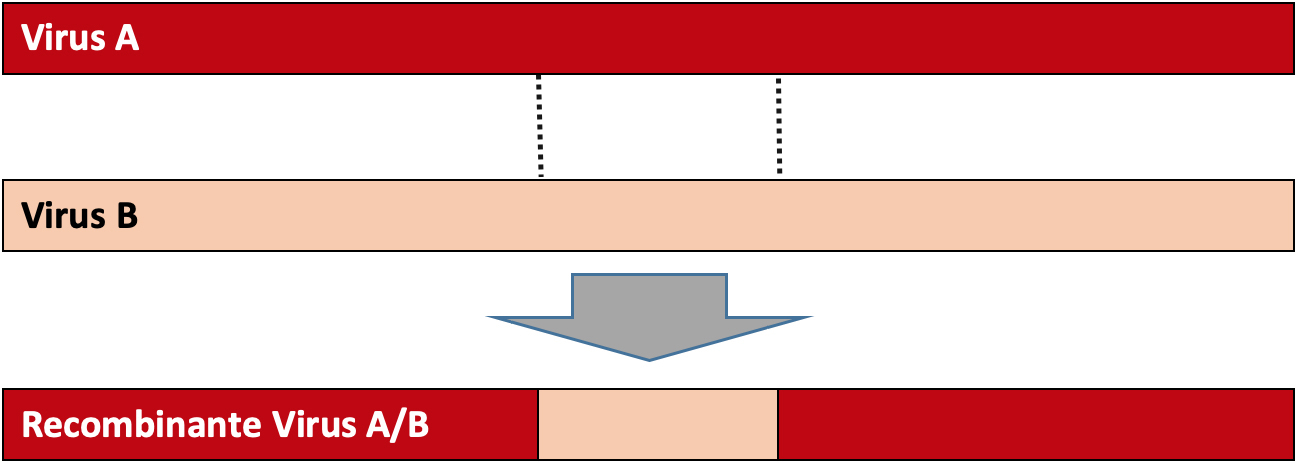
Muita controvérsia tem havido acerca da origem do SARS-CoV2, com alguns governos a acusarem os laboratórios chineses de criarem o vírus. Até ao momento, o que se sabe é que a estirpe do vírus mais próximo do SARS-CoV2 foi identificado em morcegos-ferradura (genéro Rinolophus) que viviam em cavernas precisamente em Wuhan. Essa estirpe, designada por RaTG13, apresenta apenas 96% de similaridade com o SARS-CoV2[11]. Os vírus caracterizam- -se por apresentar uma elevada taxa de mutação mas, mesmo com essas taxas de mutação elevadas, 4% de divergência correspondem a mais de 20 anos de divergência. Atualmente, a hipótese mais provável é que o SARS-CoV2 seja resultado de uma recombinação natural entre diversos genomas de coronavírus8. Já anteriormente a análise da evolução de outros coronavírus, nomeadamente do SARS-CoV, revelou a ocorrência de diversos eventos de recombinação[12]. A recombinação entre vírus ocorre numa célula de um hospedeiro, que foi infetada por múltiplas estirpes de coronavírus que trocam informação genética entre si e originam um vírus recombinante (FIGURA 2). Os Coronavírus são os vírus de RNA descritos até ao momento com o maior genoma, são segmentados e por isso constituídos por diversas proteínas.
Uma delas, a proteína da espícula (Spike), que se localiza na superfície da partícula viral, tem um domínio de ligação ao recetor humano ACE2 (Enzima Conversora da Angiotensina 2) que está localizado na membrana da superfície das células. Ao longo do seu genoma o SARS-CoV2 apresenta uma maior similaridade com a estirpe detetada no morcego-ferradura, RaTG13, no entanto, no domínio de ligação ao recetor humano localizado na proteína Spike, a semelhança é muito maior com a estirpe detetada no Pangolim, havendo uma partilha completa dos seis resíduos aminoacídicos considerados chave para a ligação com o recetor do hospedeiro ACE2[13].
Uma outra caracteristica única do SARS-CoV2 é a inserção de 4 aminoácidos na junção das subunidades 1 e 2 da proteína Spike que vão criar um sítio de clivagem para uma enzima, a furina[14] (FIGURA 3).
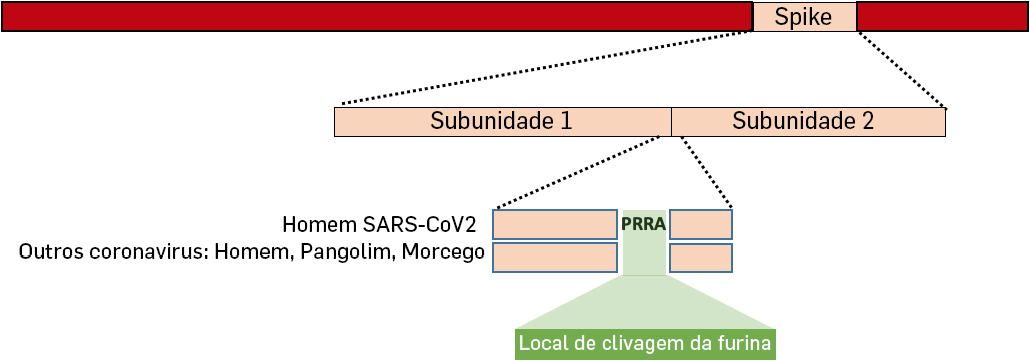
Algumas proteínas estão inativas quando são sintetizadas, e só se tornam ativas quando partes da proteína são clivadas pela furina como parece ser o caso da proteína da espícula do SARS-CoV2. Adicionalmente, esta inserção pode originar potenciais sítios de glicosilação que muito provavelmente facilitam e promovem o processo de infetividade e replicação. Uma vez que esta inserção só aparece no SARS-CoV2 e está presente em todas as sequências de SARS-CoV2 obtidas até ao momento em infeções em humanos com COVID-19, é muito provável que esta inserção seja produto de seleção natural. Esta alteração genética apareceu e provavelmente funcionou como gatilho para o ínicio da infeção em humanos tendo-se fixado. Esta seleção natural pode ter ocorrido no hospedeiro animal antes de se tornar zoonótica ou pode ter ocorrido no homem após se ter tornado zoonótica[15].
O que podemos aprender com a peste espanhola ou a peste pneumónica
Muito se tem falado e escrito sobre a evolução da COVID-19, por isso, a comparação com uma outra pandemia a peste espanhola ou pneumónica é inevitável. De facto, as semelhanças são notáveis desde as previsões de várias vagas até ás medidas propostas para conter a doença. A peste pneumónica apareceu em 1918 e foi provocada pelo vírus Influenza. Apesar de alguma controvérsia, hoje em dia aceita-se que terá tido a sua origem num campo de treino do Exército norte-americano no Kansas, USA. Tal como a COVID-19, tornou-se uma pandemia e afetou o mundo inteiro, tendo matado mais de 50 milhões de pessoas em todo o mundo e cerca de 100 mil em Portugal. Em Portugal a pneumónica deu- -se em três vagas, como no resto do mundo. A primeira ocorreu nos fins de Maio de 1918 na zona fronteiriça com Espanha e expandiu-se rapidamente para o resto do país. Teve o seu ponto máximo em Junho e não foi particularmente letal. A segunda vaga começou nos finais de Agosto na zona do Porto, mais propriamente em Vila Nova de Gaia, alastrou-se rapidamente por todo o país e foi extremamente letal. A terceira e última vaga começou em Abril de 1919, atingiu o seu pico em Outubro e foi a menos letal[16]. O Responsável pelo combate à gripe pneumómica em Portugal foi o Doutor Ricardo Jorge, Inspetor-geral de saúde na altura, que mandou implementar medidas muito semelhantes às aplicadas atualmente para a COVID-19 e foram sintetizadas por Ricardo Jorge na frase “cama, dieta, tisanas e médico”. Tal como agora, Ricardo Jorge previu que as duas únicas soluções para parar a pandemia seriam a criação de imunidade de grupo (herd immunity), em que o vírus deixa de circular quando uma percentagem muito elevada da população (sempre acima de 70%) apresentar anticorpos específicos contra o vírus, ou se fosse criada uma vacina que proteja as pessoas contra o vírus[17].
Como podemos evitar pandemias?
A probabilidade de ocorrerem novas pandemias com coronavírus, Influenza, Ébola, Zika, Dengue ou qualquer outro vírus é muitíssimo elevada. Para diminuir essa probabilidade há um conjunto de medidas que poderão e deverão ser implementadas. Entre elas destaca-se a não destruição das florestas mundiais, em particular florestas virgens ou pouco exploradas, evitando assim o contacto do Homem com novos agentes infeciosos para os quais o Homem não tem qualquer defesa. Uma nova área de investigação é caracterizar a virosphere (conjunto de todas as viroses que circulam no nosso planeta). O número de vírus caracterizados até ao momento corresponde a uma pequeníssima fração de todos os vírus existente na terra. A caracterização da virosphere é de vital importância para compreender e avaliar o risco de novas zoonoses e de infeções para a vida selvagem. Finalmente, devemos evitar ao máximo a introdução de espécies exóticas que trazem consigo novos agentes patogénicos; dois bons exemplos são a filoxera, que afetou no século XIX as videiras em França e no Douro em Portugal, provocada por um afídeo trazido para a Europa pela introdução de vinhas americanas[18], e a mixomatose, provocada pelo vírus mixoma, que no hospedeiro natural, o coelho-americano do continente americano, é benigna e que no continente europeu provoca elevadas mortalidades no coelho-bravo[19]. Duas doenças, afetando quer plantas, quer animais, com consequências devastadoras na Europa causadas pela introdução no continente europeu de espécies alóctones.
Referências
- ↑ MCINTOSH, K. Coronaviruses: A Comparative Review. Current Topics in Microbiology and Immunology / Ergebnisse der Mikrobiologie und Immunitätsforschung Springer Verlag Berlin, Heidelberg, 63, 85-129. 1974.
- ↑ KAHN, J.S. & MCINTOSH K. History and Recent Advances in Coronavirus Discovery, The Pediatric Infectious Disease Journal, 24, S223-S227. 2005.
- ↑ CORMAN, V.M., et al. Hosts and Sources of Endemic Human Coronaviruses Adv Virus Res., 100:163-188. 2018.
- ↑ CUI, J., et al. Origin and evolution of pathogenic coronaviruses, Nature Reviews Microbiology, 17: 181–192. 2019.
- ↑ WU, F., et al. A new coronavirus associated with human respiratory disease in China, Nature, volume, 579: 265–269. 2020.
- ↑ CUI, J., et al. Origin and evolution of pathogenic coronaviruses, Nature Reviews Microbiology, 17: 181–192. 2019.
- ↑ CUI, J., et al. Origin and evolution of pathogenic coronaviruses, Nature Reviews Microbiology, 17: 181–192. 2019.
- ↑ CUI, J., et al. Origin and evolution of pathogenic coronaviruses, Nature Reviews Microbiology, 17: 181–192. 2019.
- ↑ XIAO, K., et al. Isolation of SARS-CoV-2-related Coronavirus From Malayan Pangolins, Nature, Online ahead of print. 2020.
- ↑ SHI, J., et al. Susceptibility of ferrets, cats, dogs, and different domestic animals to SARS-coronavirus-2, Science, 368:1016-1020. 2020.
- ↑ ANDERSEN, K.G., et al. The proximal origin of SARS-CoV-2, Nat Med, 26, 450–452. 2020.
- ↑ GRAHAM, R.L. & BARIC, R.S. Recombination, Reservoirs, and the Modular Spike: Mechanisms of Coronavirus Cross-Species Transmission, J. Virol., 84:3134-46. 2010.
- ↑ ANDERSEN, K.G., et al. The proximal origin of SARS-CoV-2, Nat Med, 26, 450–452. 2020.
- ↑ ANDERSEN, K.G., et al. The proximal origin of SARS-CoV-2, Nat Med, 26, 450–452. 2020.
- ↑ ANDERSEN, K.G., et al. The proximal origin of SARS-CoV-2, Nat Med, 26, 450–452. 2020.
- ↑ SEQUEIRA, A. A pneumónica, Medicina Interna, 8:49-55. 2001.
- ↑ SEQUEIRA, A. A pneumónica, Medicina Interna, 8:49-55. 2001.
- ↑ SMITH, M.C. Plant Resistance to Arthropods: molecular and conventional approaches, Springer. 2005.
- ↑ FENNER, F. & RATCLIFFE, F.N. Myxomatosis, Cambridge University Press, Cambridge, 379 pp. 1965.
Recursos relacionados disponíveis na Casa das Ciências:
Criada em 24 de Junho de 2020
Revista em 2 de Julho de 2020
Aceite pelo editor em 30 de Setembro de 2020
